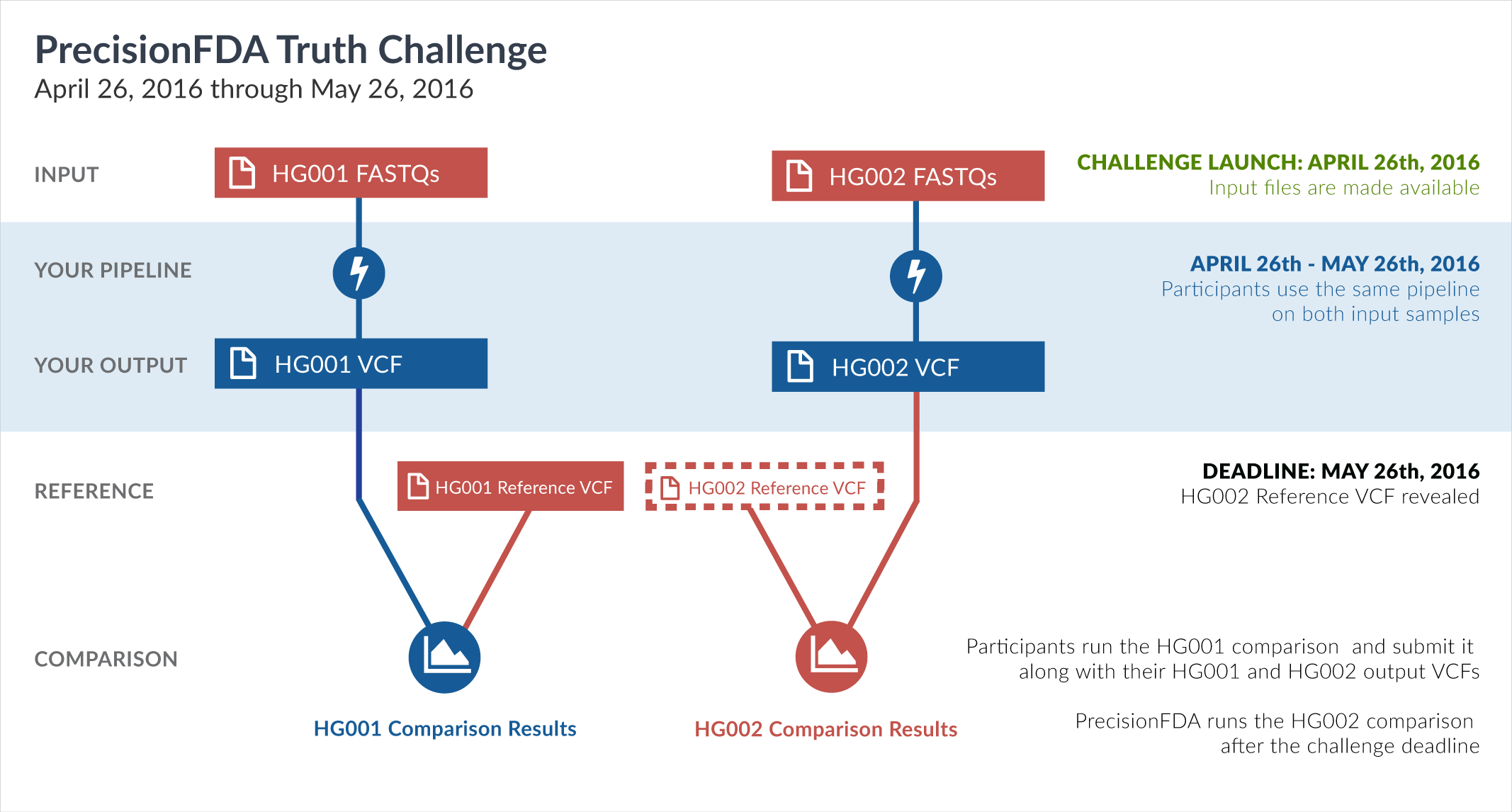
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
2301-2350 / 86044 show all | |||||||||||||||
| mlin-fermikit | INDEL | I16_PLUS | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| mlin-fermikit | INDEL | I16_PLUS | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| mlin-fermikit | INDEL | I16_PLUS | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| mlin-fermikit | INDEL | I16_PLUS | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I16_PLUS | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I16_PLUS | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I16_PLUS | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I16_PLUS | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C6_15 | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C6_15 | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C6_15 | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C6_15 | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | SNP | * | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | SNP | * | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | SNP | * | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | SNP | * | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | D6_15 | decoy | * | 100.0000 | 100.0000 | 100.0000 | 99.8596 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D6_15 | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | D6_15 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.0385 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D6_15 | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | D16_PLUS | decoy | * | 90.9091 | 83.3333 | 100.0000 | 98.3221 | 5 | 1 | 5 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D16_PLUS | decoy | het | 85.7143 | 75.0000 | 100.0000 | 98.4536 | 3 | 1 | 3 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D16_PLUS | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | D16_PLUS | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 97.2973 | 2 | 0 | 2 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D1_5 | decoy | * | 100.0000 | 100.0000 | 100.0000 | 99.8672 | 4 | 0 | 6 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D1_5 | decoy | het | 100.0000 | 100.0000 | 100.0000 | 99.8566 | 2 | 0 | 4 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D1_5 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.6364 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D1_5 | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9312 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | I1_5 | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I1_5 | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I1_5 | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I1_5 | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I6_15 | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I6_15 | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I6_15 | decoy | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | I6_15 | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | D16_PLUS | decoy | * | 90.9091 | 83.3333 | 100.0000 | 99.1482 | 5 | 1 | 5 | 0 | 0 | ||
| jpowers-varprowl | INDEL | D16_PLUS | decoy | het | 85.7143 | 75.0000 | 100.0000 | 99.4208 | 3 | 1 | 3 | 0 | 0 | ||
| jpowers-varprowl | INDEL | D16_PLUS | decoy | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | D16_PLUS | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 97.1014 | 2 | 0 | 2 | 0 | 0 | ||
| jpowers-varprowl | INDEL | D1_5 | decoy | * | 85.7143 | 75.0000 | 100.0000 | 99.9682 | 3 | 1 | 3 | 0 | 0 | ||
| jpowers-varprowl | INDEL | D1_5 | decoy | het | 100.0000 | 100.0000 | 100.0000 | 99.9748 | 2 | 0 | 2 | 0 | 0 | ||
| jpowers-varprowl | INDEL | D1_5 | decoy | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 1 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | D1_5 | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9330 | 1 | 0 | 1 | 0 | 0 | ||
| jpowers-varprowl | INDEL | * | decoy | * | 84.2105 | 80.0000 | 88.8889 | 99.9574 | 8 | 2 | 8 | 1 | 1 | 100.0000 | |
| jpowers-varprowl | INDEL | * | decoy | het | 83.3333 | 83.3333 | 83.3333 | 99.9657 | 5 | 1 | 5 | 1 | 1 | 100.0000 | |
| jpowers-varprowl | INDEL | * | decoy | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 1 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | * | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9173 | 3 | 0 | 3 | 0 | 0 | ||
| jpowers-varprowl | INDEL | I16_PLUS | decoy | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I16_PLUS | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||