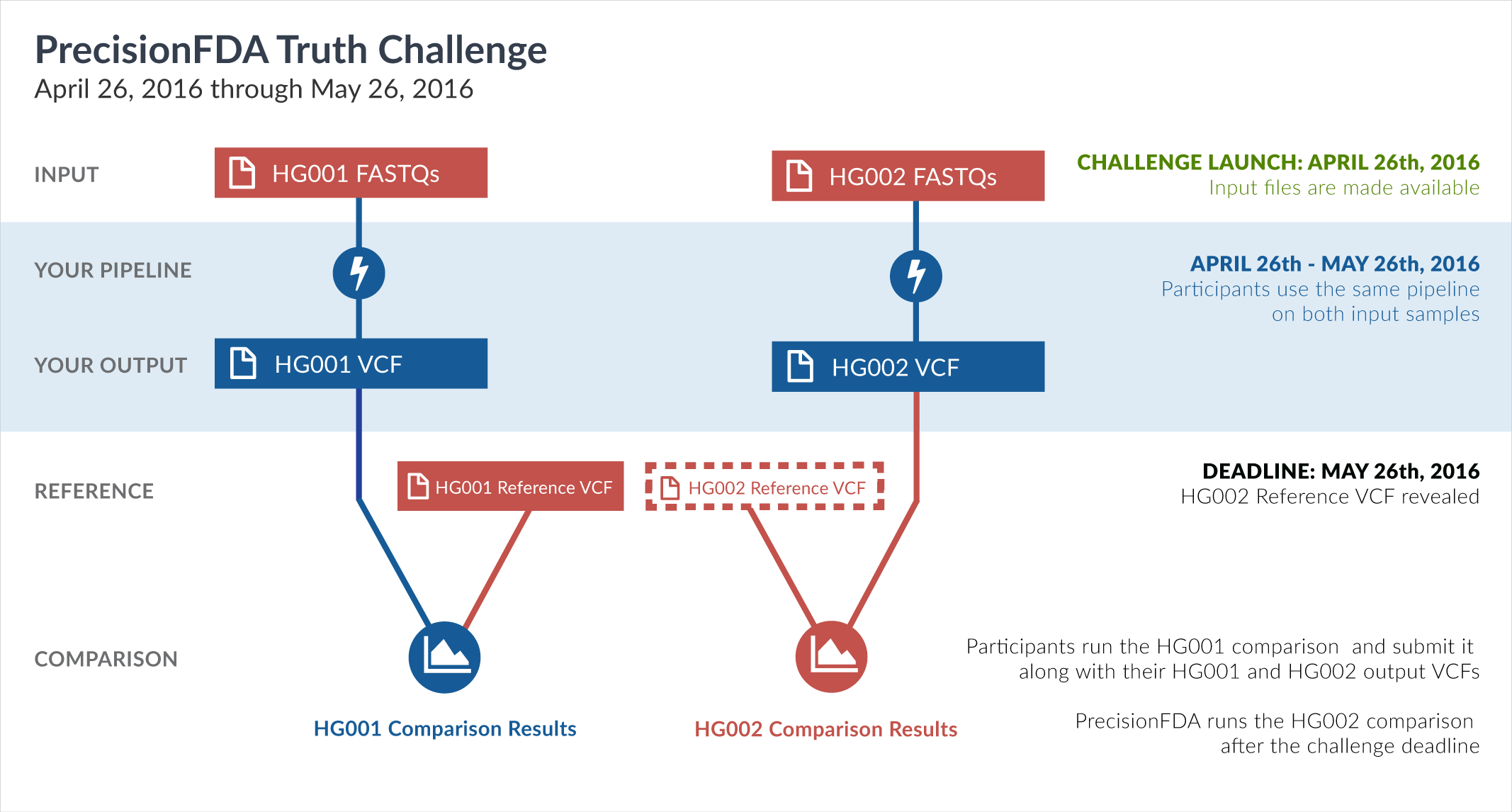
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
82901-82950 / 86044 show all | |||||||||||||||
| ltrigg-rtg1 | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9964 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9933 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | * | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | * | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I6_15 | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I6_15 | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I6_15 | segdupwithalt | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | I6_15 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I16_PLUS | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I16_PLUS | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I16_PLUS | segdupwithalt | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | I16_PLUS | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | * | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | * | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | * | segdupwithalt | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | SNP | * | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C16_PLUS | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C16_PLUS | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C16_PLUS | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ltrigg-rtg1 | INDEL | C16_PLUS | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | ti | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | ti | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | SNP | ti | segdupwithalt | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | SNP | ti | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I1_5 | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I1_5 | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jpowers-varprowl | INDEL | I1_5 | segdupwithalt | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| jpowers-varprowl | INDEL | I1_5 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I1_5 | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I1_5 | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I1_5 | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I1_5 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D1_5 | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9960 | 1 | 0 | 1 | 0 | 0 | ||
| jmaeng-gatk | INDEL | D1_5 | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9948 | 1 | 0 | 1 | 0 | 0 | ||
| jmaeng-gatk | INDEL | D1_5 | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D1_5 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D6_15 | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D6_15 | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D6_15 | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | D6_15 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I16_PLUS | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I16_PLUS | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I16_PLUS | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | INDEL | I16_PLUS | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | * | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | * | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | * | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | * | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | ti | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| jmaeng-gatk | SNP | ti | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||