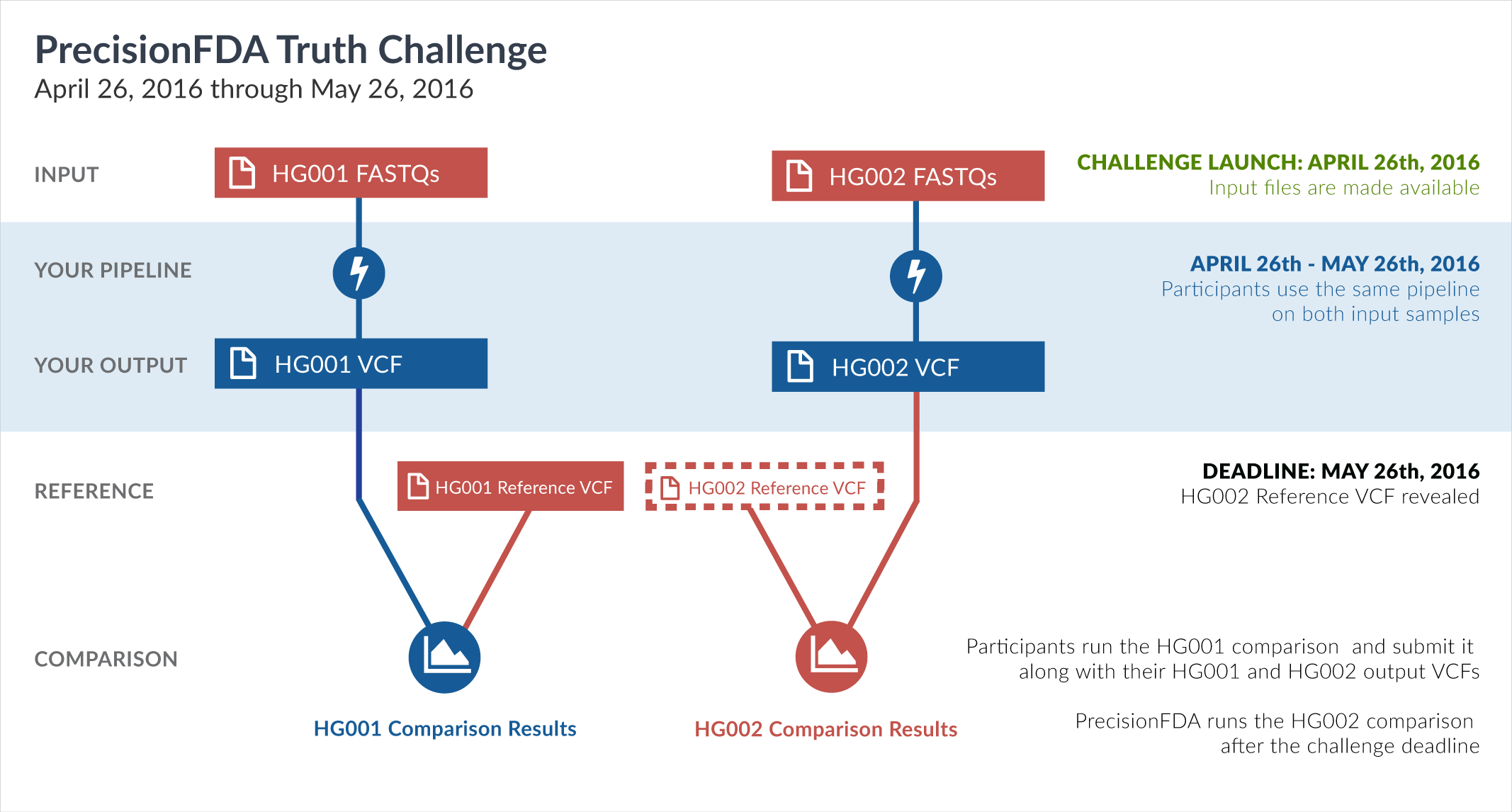
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
77001-77050 / 86044 show all | |||||||||||||||
| ckim-vqsr | INDEL | D1_5 | map_l150_m1_e0 | hetalt | 72.7273 | 57.1429 | 100.0000 | 98.3607 | 4 | 3 | 4 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l150_m2_e0 | hetalt | 72.7273 | 57.1429 | 100.0000 | 98.5507 | 4 | 3 | 4 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l150_m2_e1 | hetalt | 76.9231 | 62.5000 | 100.0000 | 98.2332 | 5 | 3 | 5 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m0_e0 | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D1_5 | map_l250_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 97.5000 | 13 | 0 | 13 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m1_e0 | hetalt | 50.0000 | 33.3333 | 100.0000 | 99.2647 | 1 | 2 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m1_e0 | homalt | 99.1150 | 98.2456 | 100.0000 | 94.6919 | 56 | 1 | 56 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m2_e0 | hetalt | 50.0000 | 33.3333 | 100.0000 | 99.3902 | 1 | 2 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m2_e0 | homalt | 99.1597 | 98.3333 | 100.0000 | 95.0669 | 59 | 1 | 59 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m2_e1 | hetalt | 50.0000 | 33.3333 | 100.0000 | 99.4048 | 1 | 2 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_l250_m2_e1 | homalt | 99.1597 | 98.3333 | 100.0000 | 95.1915 | 59 | 1 | 59 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | map_siren | hetalt | 93.6709 | 88.0952 | 100.0000 | 91.2218 | 74 | 10 | 74 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | segdup | hetalt | 98.0392 | 96.1538 | 100.0000 | 95.6336 | 50 | 2 | 51 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9960 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9946 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D1_5 | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D1_5 | tech_badpromoters | * | 97.2973 | 94.7368 | 100.0000 | 48.5714 | 18 | 1 | 18 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | tech_badpromoters | het | 93.3333 | 87.5000 | 100.0000 | 58.8235 | 7 | 1 | 7 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | tech_badpromoters | hetalt | 100.0000 | 100.0000 | 100.0000 | 0.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D1_5 | tech_badpromoters | homalt | 100.0000 | 100.0000 | 100.0000 | 43.7500 | 9 | 0 | 9 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | decoy | * | 100.0000 | 100.0000 | 100.0000 | 99.9554 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | decoy | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D6_15 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.2647 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | decoy | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D6_15 | func_cds | * | 100.0000 | 100.0000 | 100.0000 | 56.5657 | 43 | 0 | 43 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | het | 100.0000 | 100.0000 | 100.0000 | 53.9683 | 29 | 0 | 29 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | hetalt | 100.0000 | 100.0000 | 100.0000 | 60.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | homalt | 100.0000 | 100.0000 | 100.0000 | 61.2903 | 12 | 0 | 12 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.1831 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 97.8873 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 89.4737 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 98.0769 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_51to200bp_gt95identity_merged | hetalt | 96.0000 | 92.3077 | 100.0000 | 56.6265 | 36 | 3 | 36 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.8723 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 98.4252 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 84.6154 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 92.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 93.3333 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 75.0000 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_6to10 | het | 100.0000 | 100.0000 | 100.0000 | 85.2381 | 155 | 0 | 155 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_6to10 | homalt | 100.0000 | 100.0000 | 100.0000 | 82.3810 | 74 | 0 | 74 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.1788 | 9 | 0 | 9 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | homalt | 100.0000 | 100.0000 | 100.0000 | 99.8102 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_quadTR_gt200 | * | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_quadTR_gt200 | het | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_quadTR_gt200 | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_quadTR_gt200 | homalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||