PrecisionFDA
Truth Challenge
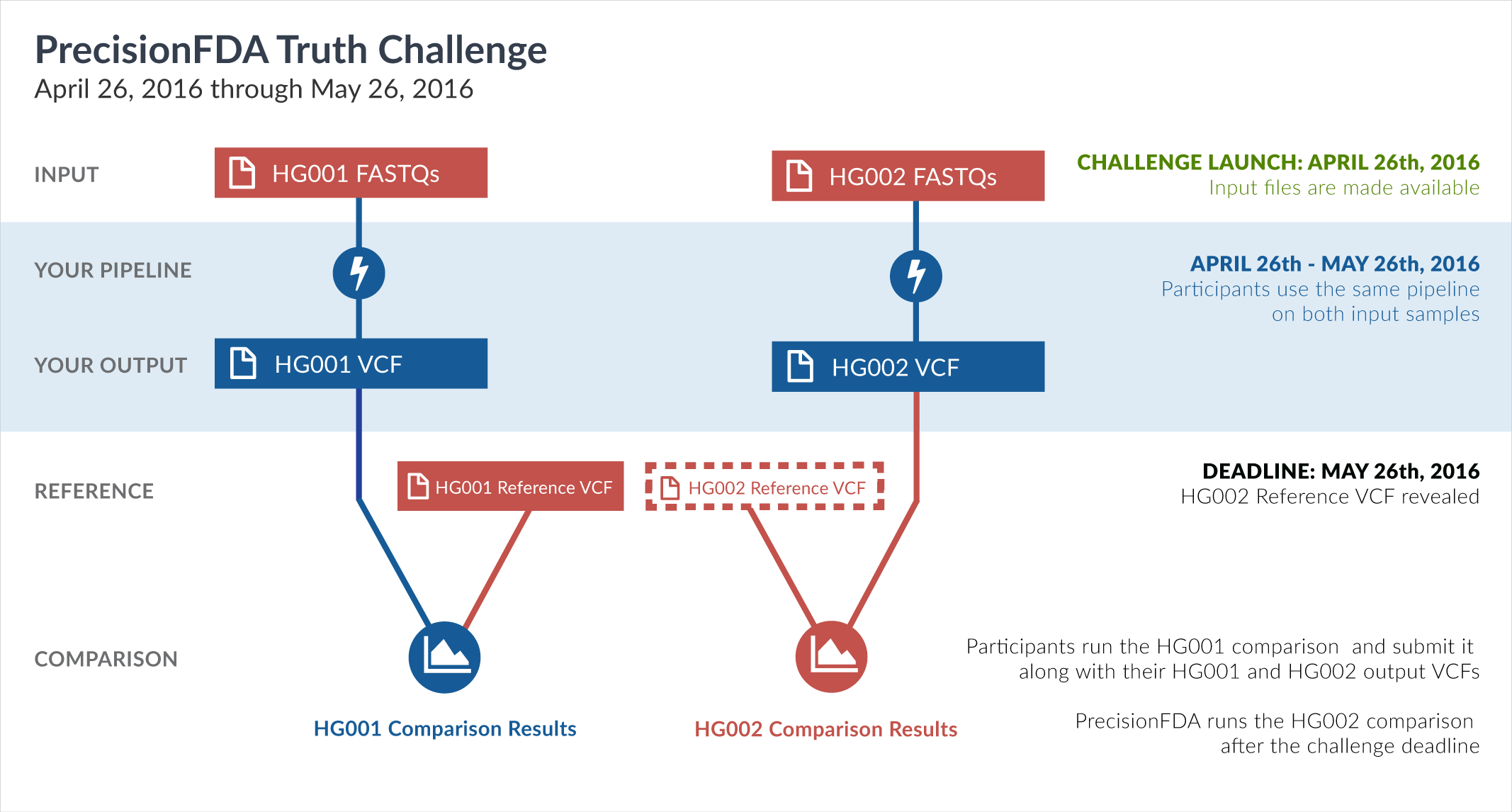
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
5151-5200 / 86044 show all | |||||||||||||||
| ckim-vqsr | INDEL | D6_15 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.2647 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | * | 100.0000 | 100.0000 | 100.0000 | 56.5657 | 43 | 0 | 43 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | het | 100.0000 | 100.0000 | 100.0000 | 53.9683 | 29 | 0 | 29 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | hetalt | 100.0000 | 100.0000 | 100.0000 | 60.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | func_cds | homalt | 100.0000 | 100.0000 | 100.0000 | 61.2903 | 12 | 0 | 12 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.1831 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 97.8873 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 89.4737 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 98.0769 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_51to200bp_gt95identity_merged | homalt | 98.8506 | 100.0000 | 97.7273 | 77.7778 | 43 | 0 | 43 | 1 | 1 | 100.0000 | |
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.8723 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 98.4252 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 84.6154 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt101bp_gt95identity_merged | homalt | 99.8665 | 100.0000 | 99.7333 | 59.5032 | 374 | 0 | 374 | 1 | 0 | 0.0000 | |
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt51bp_gt95identity_merged | het | 99.8974 | 100.0000 | 99.7951 | 68.0419 | 487 | 0 | 487 | 1 | 1 | 100.0000 | |
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt51bp_gt95identity_merged | homalt | 99.8544 | 100.0000 | 99.7093 | 57.2671 | 343 | 0 | 343 | 1 | 0 | 0.0000 | |
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 92.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 93.3333 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 75.0000 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_6to10 | het | 100.0000 | 100.0000 | 100.0000 | 85.2381 | 155 | 0 | 155 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_6to10 | homalt | 100.0000 | 100.0000 | 100.0000 | 82.3810 | 74 | 0 | 74 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.1788 | 9 | 0 | 9 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | homalt | 100.0000 | 100.0000 | 100.0000 | 99.8102 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_quadTR_51to200 | homalt | 98.7147 | 100.0000 | 97.4619 | 47.3262 | 192 | 0 | 192 | 5 | 4 | 80.0000 | |
| ckim-vqsr | INDEL | D6_15 | lowcmp_SimpleRepeat_triTR_51to200 | homalt | 100.0000 | 100.0000 | 100.0000 | 36.3636 | 21 | 0 | 21 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l125_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 90.9091 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l125_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 93.1429 | 12 | 0 | 12 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l150_m0_e0 | * | 96.9697 | 100.0000 | 94.1176 | 95.6242 | 32 | 0 | 32 | 2 | 0 | 0.0000 | |
| ckim-vqsr | INDEL | D6_15 | map_l150_m0_e0 | het | 95.2381 | 100.0000 | 90.9091 | 96.1938 | 20 | 0 | 20 | 2 | 0 | 0.0000 | |
| ckim-vqsr | INDEL | D6_15 | map_l150_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 91.2281 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l150_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 95.0704 | 7 | 0 | 7 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l150_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 91.8367 | 8 | 0 | 8 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l150_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 92.7928 | 8 | 0 | 8 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m0_e0 | * | 100.0000 | 100.0000 | 100.0000 | 98.5782 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m0_e0 | het | 100.0000 | 100.0000 | 100.0000 | 98.7179 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 97.4684 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m1_e0 | * | 100.0000 | 100.0000 | 100.0000 | 97.7011 | 18 | 0 | 18 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m1_e0 | het | 100.0000 | 100.0000 | 100.0000 | 98.1034 | 11 | 0 | 11 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 96.6667 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m1_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 96.5035 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e0 | * | 100.0000 | 100.0000 | 100.0000 | 97.5637 | 22 | 0 | 22 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e0 | het | 100.0000 | 100.0000 | 100.0000 | 97.8979 | 14 | 0 | 14 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 97.2973 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 96.3190 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e1 | * | 100.0000 | 100.0000 | 100.0000 | 97.6293 | 22 | 0 | 22 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e1 | het | 100.0000 | 100.0000 | 100.0000 | 97.9622 | 14 | 0 | 14 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 97.3333 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | map_l250_m2_e1 | homalt | 100.0000 | 100.0000 | 100.0000 | 96.3855 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-vqsr | INDEL | D6_15 | segdup | homalt | 96.1538 | 100.0000 | 92.5926 | 92.3944 | 50 | 0 | 50 | 4 | 4 | 100.0000 | |
| ckim-vqsr | INDEL | D6_15 | tech_badpromoters | * | 100.0000 | 100.0000 | 100.0000 | 51.4286 | 17 | 0 | 17 | 0 | 0 | ||