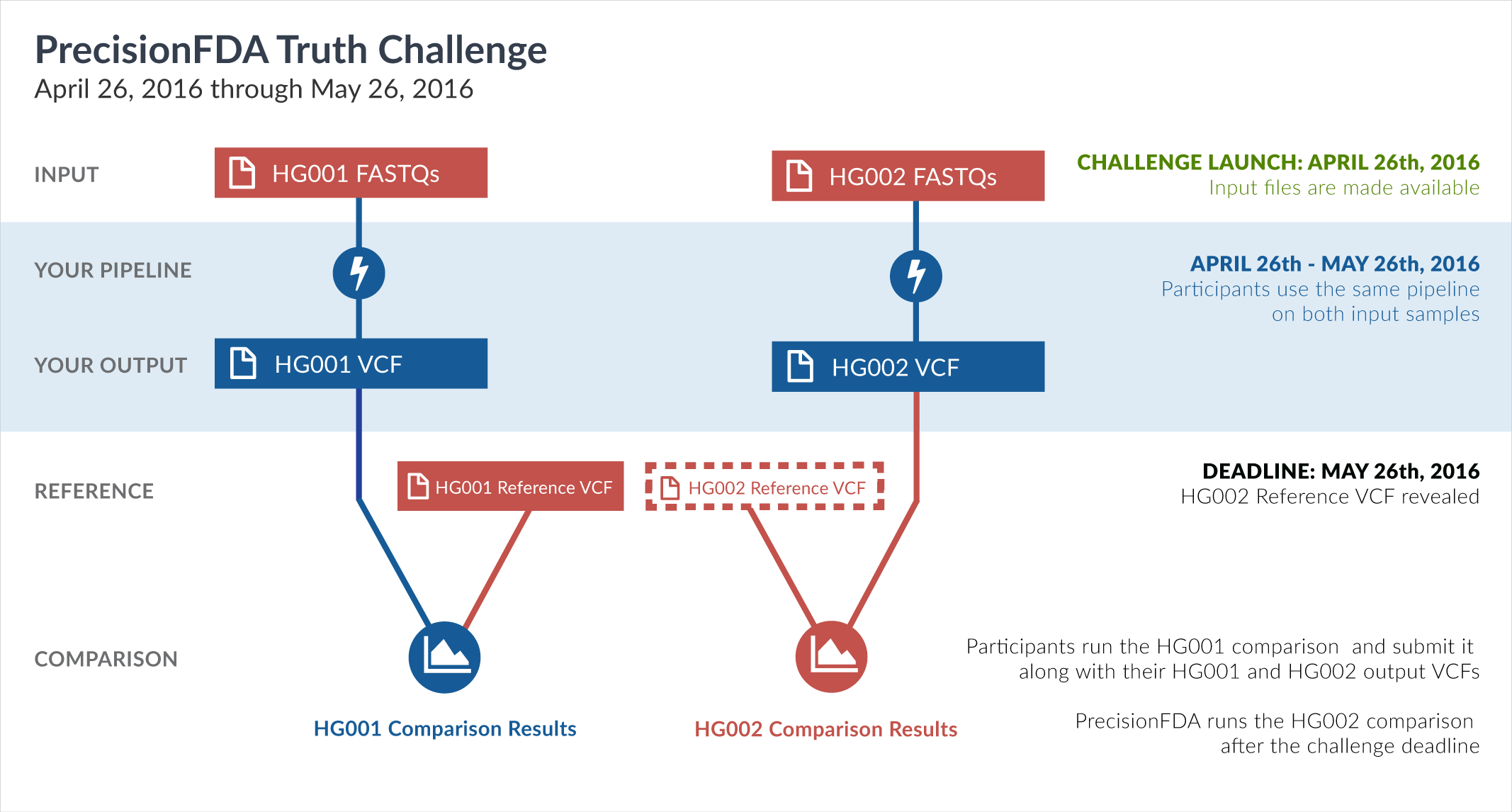
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
75101-75150 / 86044 show all | |||||||||||||||
| jlack-gatk | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 40.0000 | 100.0000 | 25.0000 | 99.9965 | 1 | 0 | 1 | 3 | 0 | 0.0000 | |
| gduggal-snapplat | INDEL | D1_5 | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9965 | 1 | 0 | 1 | 0 | 0 | ||
| asubramanian-gatk | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 100.0000 | 100.0000 | 100.0000 | 99.9969 | 1 | 0 | 4 | 0 | 0 | ||
| raldana-dualsentieon | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9969 | 1 | 0 | 1 | 0 | 0 | ||
| qzeng-custom | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9969 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9970 | 1 | 0 | 1 | 0 | 0 | ||
| hfeng-pmm3 | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9970 | 1 | 0 | 1 | 0 | 0 | ||
| jli-custom | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9971 | 1 | 0 | 1 | 0 | 0 | ||
| rpoplin-dv42 | INDEL | * | decoy | * | 94.7368 | 90.0000 | 100.0000 | 99.9971 | 9 | 1 | 9 | 0 | 0 | ||
| hfeng-pmm1 | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9971 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-snapfb | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9971 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-bwafb | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9972 | 1 | 0 | 1 | 0 | 0 | ||
| ndellapenna-hhga | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 50.0000 | 100.0000 | 33.3333 | 99.9972 | 1 | 0 | 1 | 2 | 0 | 0.0000 | |
| egarrison-hhga | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 50.0000 | 100.0000 | 33.3333 | 99.9972 | 1 | 0 | 1 | 2 | 0 | 0.0000 | |
| jpowers-varprowl | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9972 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-bwavard | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-bwaplat | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| hfeng-pmm2 | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| mlin-fermikit | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 50.0000 | 100.0000 | 33.3333 | 99.9973 | 1 | 0 | 1 | 2 | 2 | 100.0000 | |
| ghariani-varprowl | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| ciseli-custom | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| ciseli-custom | SNP | * | decoy | homalt | 0.0000 | 0.0000 | 100.0000 | 99.9973 | 0 | 0 | 1 | 0 | 0 | ||
| anovak-vg | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9973 | 1 | 0 | 1 | 0 | 0 | ||
| cchapple-custom | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9974 | 1 | 0 | 1 | 0 | 0 | ||
| anovak-vg | SNP | * | decoy | homalt | 0.0000 | 0.0000 | 100.0000 | 99.9974 | 0 | 0 | 1 | 0 | 0 | ||
| astatham-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9974 | 1 | 0 | 1 | 0 | 0 | ||
| bgallagher-sentieon | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9974 | 1 | 0 | 1 | 0 | 0 | ||
| jlack-gatk | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| jmaeng-gatk | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| dgrover-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-gatk | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-snapplat | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-snapvard | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9976 | 1 | 0 | 1 | 0 | 0 | ||
| qzeng-custom | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9977 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-bwavard | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9978 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-snapvard | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9979 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9979 | 1 | 0 | 1 | 0 | 0 | ||
| eyeh-varpipe | SNP | ti | segdupwithalt | homalt | 0.0000 | 0.0000 | 100.0000 | 99.9980 | 0 | 0 | 1 | 0 | 0 | ||
| gduggal-bwaplat | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9980 | 1 | 0 | 1 | 0 | 0 | ||
| jlack-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 1 | 0 | 0 | ||
| ltrigg-rtg2 | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 2 | 0 | 0 | ||
| ltrigg-rtg1 | INDEL | D1_5 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 2 | 0 | 0 | ||
| ckim-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-vqsr | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 1 | 0 | 0 | ||
| gduggal-snapplat | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9982 | 1 | 0 | 1 | 0 | 0 | ||
| jmaeng-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9982 | 1 | 0 | 1 | 0 | 0 | ||
| asubramanian-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9985 | 1 | 0 | 2 | 0 | 0 | ||
| anovak-vg | SNP | ti | decoy | * | 0.0000 | 0.0000 | 50.0000 | 99.9986 | 0 | 0 | 1 | 1 | 1 | 100.0000 | |
| anovak-vg | SNP | tv | decoy | het | 0.0000 | 0.0000 | 99.9986 | 0 | 0 | 0 | 1 | 1 | 100.0000 | ||