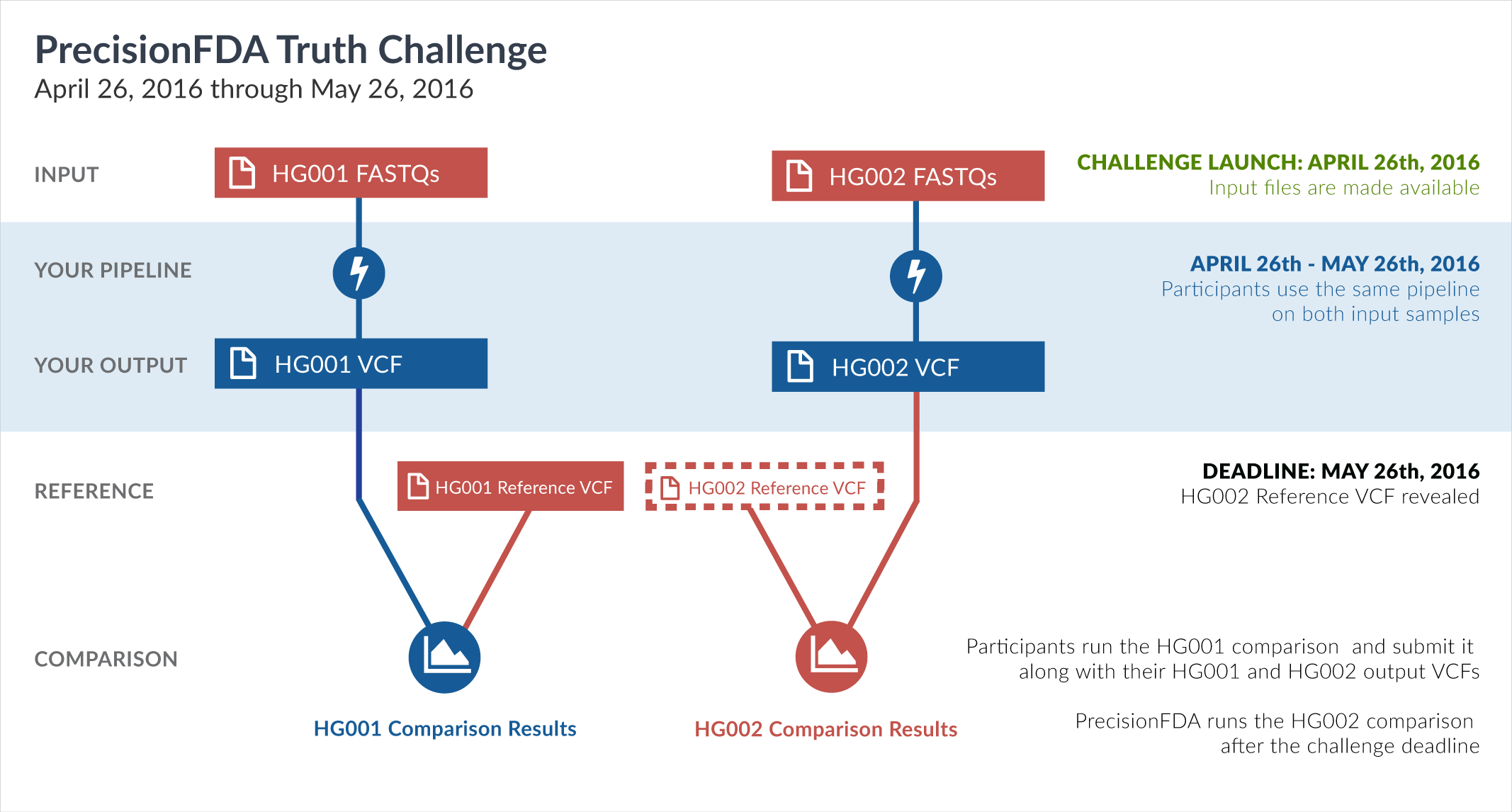
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
82401-82450 / 86044 show all | |||||||||||||||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 87.5000 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt101bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 70.8861 | 19 | 0 | 23 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt51bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 41.6667 | 12 | 0 | 14 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_SimpleRepeat_homopolymer_6to10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 52.9412 | 12 | 0 | 16 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_SimpleRepeat_homopolymer_6to10 | homalt | 100.0000 | 100.0000 | 100.0000 | 87.2928 | 23 | 0 | 23 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_SimpleRepeat_triTR_11to50 | hetalt | 100.0000 | 100.0000 | 100.0000 | 23.9437 | 51 | 0 | 54 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | lowcmp_SimpleRepeat_triTR_11to50 | homalt | 100.0000 | 100.0000 | 100.0000 | 68.9655 | 45 | 0 | 45 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | map_l125_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 98.5612 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | map_l250_m2_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 98.7952 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | map_l250_m2_e1 | homalt | 100.0000 | 100.0000 | 100.0000 | 98.8095 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | tech_badpromoters | * | 100.0000 | 100.0000 | 100.0000 | 42.8571 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | INDEL | D16_PLUS | tech_badpromoters | het | 100.0000 | 100.0000 | 100.0000 | 0.0000 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | decoy | * | 100.0000 | 100.0000 | 100.0000 | 99.9714 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | decoy | het | 100.0000 | 100.0000 | 100.0000 | 99.9833 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.6610 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9407 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | func_cds | homalt | 100.0000 | 100.0000 | 100.0000 | 29.5238 | 74 | 0 | 74 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 98.8197 | 11 | 0 | 11 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 98.6949 | 8 | 0 | 8 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 96.8750 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 99.3031 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 98.8610 | 10 | 0 | 10 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 98.7805 | 7 | 0 | 7 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 96.5517 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 99.2727 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 98.1481 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 97.4359 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | map_l250_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 97.0252 | 13 | 0 | 13 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9953 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9935 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | tech_badpromoters | hetalt | 100.0000 | 100.0000 | 100.0000 | 0.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D1_5 | tech_badpromoters | homalt | 100.0000 | 100.0000 | 100.0000 | 47.0588 | 9 | 0 | 9 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | decoy | * | 100.0000 | 100.0000 | 100.0000 | 99.9525 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.0654 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | func_cds | hetalt | 100.0000 | 100.0000 | 100.0000 | 60.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | func_cds | homalt | 100.0000 | 100.0000 | 100.0000 | 60.0000 | 12 | 0 | 12 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.2973 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 97.9730 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 90.4762 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_AllRepeats_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 98.1132 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.9899 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 98.5185 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 86.6667 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 91.3043 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 92.3077 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | INDEL | D6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 75.0000 | 1 | 0 | 1 | 0 | 0 | ||
| cchapple-custom | INDEL | I6_15 | lowcmp_SimpleRepeat_triTR_51to200 | het | 100.0000 | 100.0000 | 100.0000 | 61.2903 | 1 | 0 | 12 | 0 | 0 | ||
| cchapple-custom | INDEL | I6_15 | map_l125_m0_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 91.4286 | 6 | 0 | 6 | 0 | 0 | ||
| cchapple-custom | INDEL | I6_15 | map_l125_m1_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 89.5105 | 15 | 0 | 15 | 0 | 0 | ||
| cchapple-custom | INDEL | I6_15 | map_l125_m2_e0 | homalt | 100.0000 | 100.0000 | 100.0000 | 90.7975 | 15 | 0 | 15 | 0 | 0 | ||