PrecisionFDA
Truth Challenge
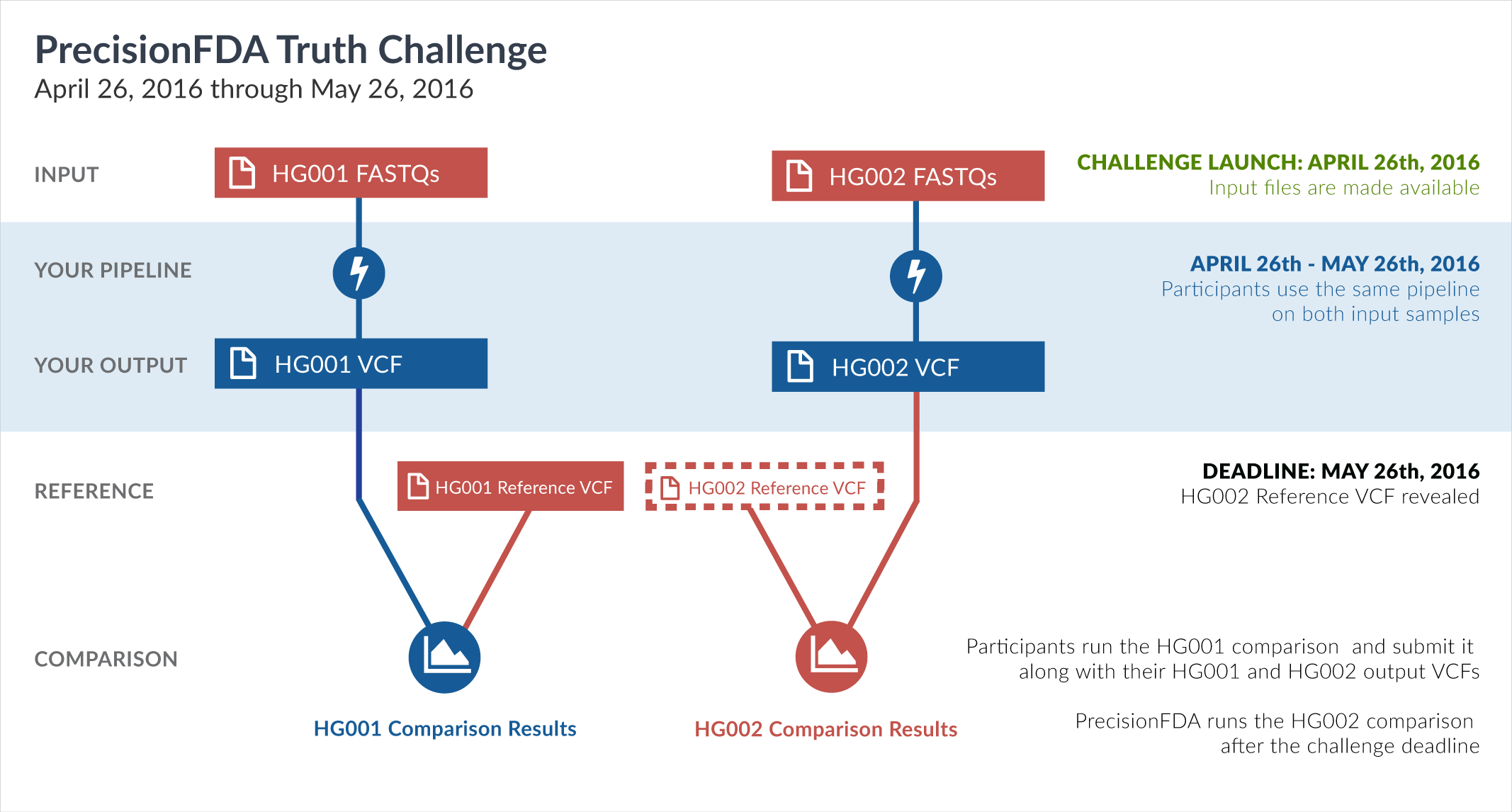
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
82151-82200 / 86044 show all | |||||||||||||||
| ckim-dragen | SNP | ti | map_l100_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 77.3723 | 31 | 0 | 31 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l125_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 81.3953 | 8 | 0 | 8 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l125_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 75.0000 | 24 | 0 | 24 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l125_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 78.5714 | 24 | 0 | 24 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l125_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 78.5714 | 24 | 0 | 24 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l150_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 91.4286 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l150_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 80.7692 | 15 | 0 | 15 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l150_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 83.1461 | 15 | 0 | 15 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l150_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 83.1461 | 15 | 0 | 15 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l250_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 91.4894 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l250_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 90.5660 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-dragen | SNP | ti | map_l250_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 90.5660 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-dragen | SNP | tv | func_cds | hetalt | 100.0000 | 100.0000 | 100.0000 | 64.2857 | 10 | 0 | 10 | 0 | 0 | ||
| ckim-dragen | SNP | tv | func_cds | homalt | 100.0000 | 100.0000 | 100.0000 | 26.6781 | 1704 | 0 | 1704 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt101bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 88.8889 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt51bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 83.3333 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_lt101bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 90.0000 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_lt51bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 88.8889 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_Human_Full_Genome_TRDB_hg19_150331_all_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 92.8571 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_diTR_11to50 | hetalt | 100.0000 | 100.0000 | 100.0000 | 95.2381 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_homopolymer_6to10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 70.5882 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_quadTR_11to50 | hetalt | 100.0000 | 100.0000 | 100.0000 | 66.6667 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_quadTR_51to200 | homalt | 100.0000 | 100.0000 | 100.0000 | 93.2584 | 6 | 0 | 6 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | * | 100.0000 | 100.0000 | 100.0000 | 98.6111 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | het | 100.0000 | 100.0000 | 100.0000 | 98.3333 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l100_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 79.4872 | 16 | 0 | 16 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l125_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 85.9375 | 9 | 0 | 9 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l150_m0_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 94.5455 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l250_m1_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 94.2857 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l250_m2_e0 | hetalt | 100.0000 | 100.0000 | 100.0000 | 93.8272 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-dragen | SNP | tv | map_l250_m2_e1 | hetalt | 100.0000 | 100.0000 | 100.0000 | 93.8272 | 5 | 0 | 5 | 0 | 0 | ||
| ckim-gatk | INDEL | * | decoy | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.8485 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-gatk | INDEL | * | decoy | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9337 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_AllRepeats_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 99.4426 | 12 | 0 | 12 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_AllRepeats_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 95.3846 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 99.5157 | 10 | 0 | 10 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_gt200bp_gt95identity_merged | hetalt | 100.0000 | 100.0000 | 100.0000 | 94.5455 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | * | 100.0000 | 100.0000 | 100.0000 | 97.5806 | 3 | 0 | 3 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | het | 100.0000 | 100.0000 | 100.0000 | 97.7273 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRlt7_gt200bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 96.1538 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_SimpleRepeat_homopolymer_gt10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.8797 | 16 | 0 | 16 | 0 | 0 | ||
| ckim-gatk | INDEL | * | lowcmp_SimpleRepeat_homopolymer_gt10 | homalt | 100.0000 | 100.0000 | 100.0000 | 99.9634 | 21 | 0 | 21 | 0 | 0 | ||
| ckim-gatk | INDEL | * | segdupwithalt | * | 100.0000 | 100.0000 | 100.0000 | 99.9981 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-gatk | INDEL | * | segdupwithalt | het | 100.0000 | 100.0000 | 100.0000 | 99.9975 | 1 | 0 | 1 | 0 | 0 | ||
| ckim-gatk | INDEL | * | tech_badpromoters | hetalt | 100.0000 | 100.0000 | 100.0000 | 50.0000 | 4 | 0 | 4 | 0 | 0 | ||
| ckim-gatk | INDEL | * | tech_badpromoters | homalt | 100.0000 | 100.0000 | 100.0000 | 57.1429 | 33 | 0 | 33 | 0 | 0 | ||
| ckim-gatk | INDEL | I6_15 | lowcmp_Human_Full_Genome_TRDB_hg19_150331_TRgt6_lt51bp_gt95identity_merged | homalt | 100.0000 | 100.0000 | 100.0000 | 70.6897 | 68 | 0 | 68 | 0 | 0 | ||
| ckim-gatk | INDEL | I6_15 | lowcmp_SimpleRepeat_homopolymer_6to10 | homalt | 100.0000 | 100.0000 | 100.0000 | 81.5789 | 49 | 0 | 49 | 0 | 0 | ||
| ckim-gatk | INDEL | I6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 100.0000 | 100.0000 | 100.0000 | 99.8656 | 2 | 0 | 2 | 0 | 0 | ||
| ckim-gatk | INDEL | I6_15 | lowcmp_SimpleRepeat_homopolymer_gt10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 99.3243 | 2 | 0 | 2 | 0 | 0 | ||