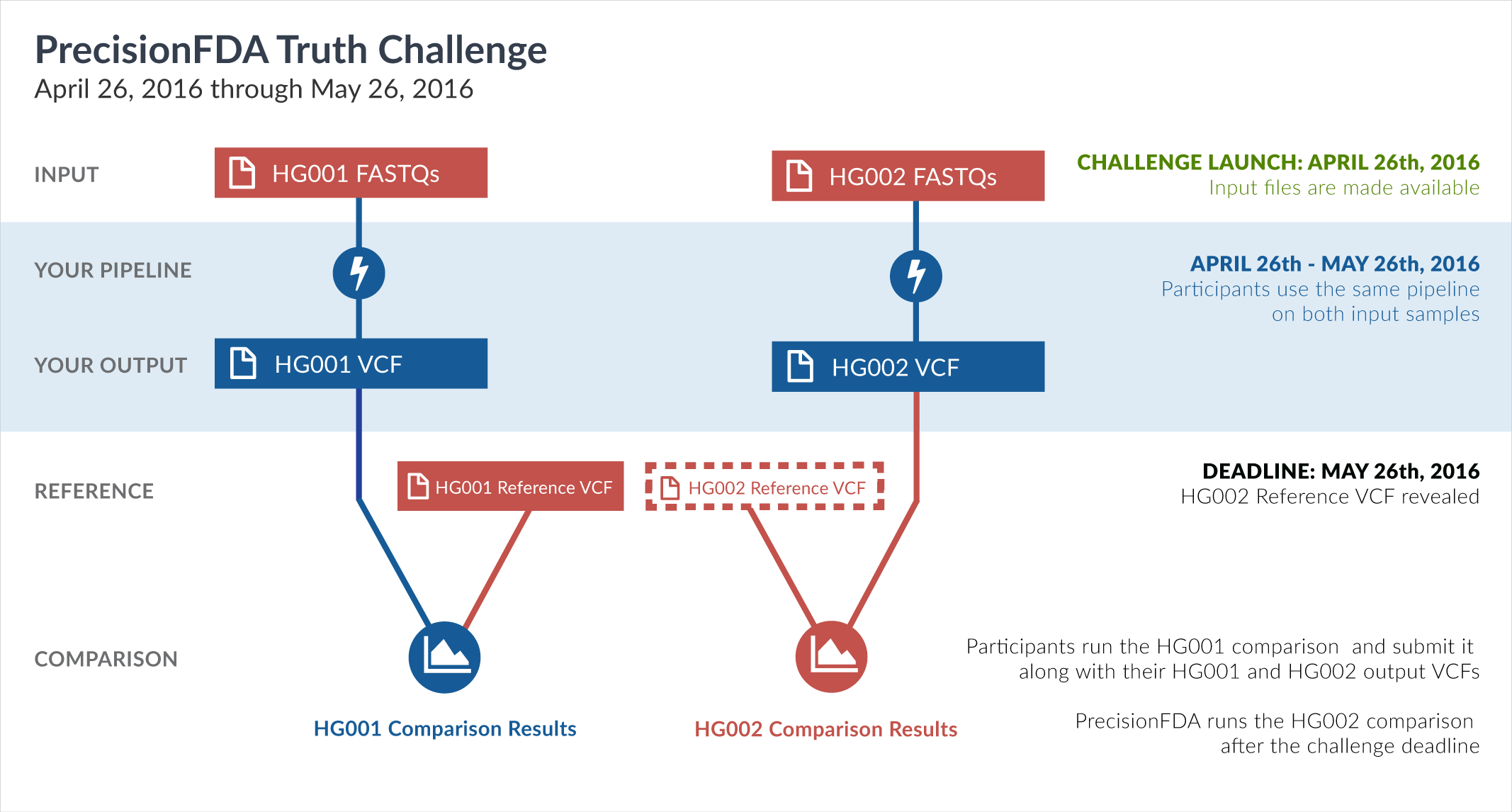
PrecisionFDA
Truth Challenge
Engage and improve DNA test results with our community challenges
Explore HG002 comparison results
Use this interactive explorer to filter all results across submission entries and multiple dimensions.
| Entry | Type | Subtype | Subset | Genotype | F-score | Recall | Precision | Frac_NA | Truth TP | Truth FN | Query TP | Query FP | FP gt | % FP ma | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
68401-68450 / 86044 show all | |||||||||||||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_diTR_gt200 | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_diTR_gt200 | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_diTR_gt200 | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_6to10 | hetalt | 100.0000 | 100.0000 | 100.0000 | 76.1905 | 5 | 0 | 5 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_6to10 | homalt | 91.6132 | 84.5897 | 99.9088 | 64.9616 | 3288 | 599 | 3285 | 3 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_gt10 | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_gt10 | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_gt10 | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_homopolymer_gt10 | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_11to50 | hetalt | 88.8889 | 80.0000 | 100.0000 | 71.4286 | 4 | 1 | 4 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_51to200 | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_51to200 | homalt | 20.0000 | 16.6667 | 25.0000 | 97.2973 | 1 | 5 | 1 | 3 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_gt200 | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_gt200 | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_gt200 | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_quadTR_gt200 | homalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_11to50 | hetalt | 0.0000 | 100.0000 | 0 | 1 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | * | 0.0000 | 0.0000 | 92.2078 | 0 | 1 | 0 | 18 | 0 | 0.0000 | ||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | het | 0.0000 | 0.0000 | 91.2621 | 0 | 1 | 0 | 18 | 0 | 0.0000 | ||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_51to200 | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_gt200 | * | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_gt200 | het | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_gt200 | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | lowcmp_SimpleRepeat_triTR_gt200 | homalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | map_l100_m0_e0 | homalt | 92.5848 | 86.1934 | 100.0000 | 67.9954 | 3315 | 531 | 3316 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l125_m0_e0 | homalt | 90.2668 | 82.2602 | 100.0000 | 75.6753 | 1827 | 394 | 1828 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l125_m1_e0 | homalt | 93.4557 | 87.7304 | 99.9806 | 69.1888 | 5141 | 719 | 5142 | 1 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | map_l125_m2_e0 | homalt | 93.5527 | 87.9009 | 99.9811 | 71.5032 | 5289 | 728 | 5290 | 1 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | map_l125_m2_e1 | homalt | 93.5797 | 87.9486 | 99.9813 | 71.5146 | 5342 | 732 | 5342 | 1 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | map_l150_m0_e0 | homalt | 88.6840 | 79.6687 | 100.0000 | 81.7822 | 1058 | 270 | 1059 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l150_m1_e0 | homalt | 91.9496 | 85.0988 | 100.0000 | 74.0535 | 3358 | 588 | 3358 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l150_m2_e0 | homalt | 92.1411 | 85.4274 | 100.0000 | 76.0916 | 3488 | 595 | 3488 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l150_m2_e1 | homalt | 92.2034 | 85.5346 | 100.0000 | 76.0404 | 3536 | 598 | 3535 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l250_m0_e0 | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | map_l250_m0_e0 | homalt | 85.4599 | 74.6114 | 100.0000 | 95.3201 | 144 | 49 | 144 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l250_m1_e0 | hetalt | 85.7143 | 75.0000 | 100.0000 | 97.0000 | 3 | 1 | 3 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l250_m1_e0 | homalt | 86.4721 | 76.1682 | 100.0000 | 89.7193 | 652 | 204 | 652 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l250_m2_e0 | homalt | 87.3121 | 77.4813 | 100.0000 | 90.2170 | 726 | 211 | 726 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | map_l250_m2_e1 | homalt | 87.3139 | 77.4841 | 100.0000 | 90.2591 | 733 | 213 | 733 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | segdup | hetalt | 100.0000 | 100.0000 | 100.0000 | 97.8125 | 7 | 0 | 7 | 0 | 0 | ||
| gduggal-snapplat | SNP | tv | segdupwithalt | * | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | segdupwithalt | het | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | segdupwithalt | hetalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | segdupwithalt | homalt | 0.0000 | 100.0000 | 0 | 0 | 0 | 0 | 0 | ||||
| gduggal-snapplat | SNP | tv | tech_badpromoters | * | 89.0511 | 84.7222 | 93.8462 | 77.3519 | 61 | 11 | 61 | 4 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | tech_badpromoters | het | 87.8788 | 87.8788 | 87.8788 | 83.6634 | 29 | 4 | 29 | 4 | 0 | 0.0000 | |
| gduggal-snapplat | SNP | tv | tech_badpromoters | hetalt | 0.0000 | 0.0000 | 0.0000 | 0 | 0 | 0 | 0 | 0 | |||
| gduggal-snapplat | SNP | tv | tech_badpromoters | homalt | 90.1408 | 82.0513 | 100.0000 | 62.3529 | 32 | 7 | 32 | 0 | 0 | ||
| gduggal-snapvard | INDEL | * | * | hetalt | 0.0000 | 37.0438 | 0.0000 | 0.0000 | 9348 | 15887 | 0 | 0 | 0 | ||